La phase 1
La pahse 1, lancé au printemps 2021, proposée par le CATI IMOTEP et ses participants visait à proposer une preuve de concept par :
- une étude de faisabilité
- l’identification des acteurs du projet
- l’identification des technologies et outils mis en oeuvres
- la réalisation d’une version simple utilisable (la version Bêta
Cette phase se voulait être une première brique répondant à la satisfaction des besoins des différentes plateformes d’épidémiosurveillances pour leurs applications.
Propositions
Des solutions libres et opensources sont apparues ces dernières années dans la communauté R.
Au vu de ces différentes possibilités et le background des membres du projet sur cette thématique, une solution a rapidement émergé.
Le projet dans sa phase 1 vise à offrir un service basé sur GitLab pour la gestion du code, la création et le stockage des images (docker) des applications et de déployer automatiquement les applications sur un cluster Kubernetes (K8S) fourni par la DSI INRAE.
Le but est de fournir un hébergement d’une application R-Shiny accèssible via une URL public et d’accompagner et simplifier ce processus pour les utilisateurs.
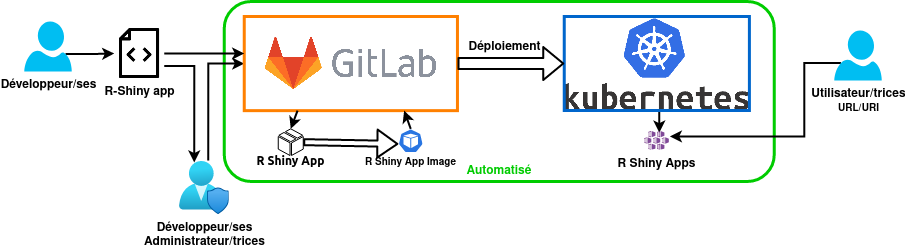
Une partie d’une telle infrastructure est déjà en production au sein de l’unité BioSP pour la Plateforme d’Epidémiosurveillance en Santé Végétale.
- Les sources de cette solution : https://gitlab.paca.inrae.fr/r-ecosystem/cookbooks/r-shiny-app-ci-cd.
- Des explications : https://gitlab.paca.inrae.fr/r-ecosystem/docs/jdev2020.
- Plus récemment : https://gitlab.paca.inrae.fr/r-ecosystem/docs/rr2021.
Organisation
Le projet SK8 regroupe plusieurs acteurs INRAE et quelques extérieurs qui participent à différents groupes de travail.
Il est principalement composé des agents du CATI IMOTEP, issus des Départements MathNUM, SPE et SA. Plusieurs membres sont issus des différentes plateformes d’épidémiosurveillance PESV, PESA et PSCA.
Le projet compte aussi la participation d’agents de la DipSO et de la DSI INRAE.
Plusieurs agents INRAE et extérieurs sont aussi issus de différentes UMRs.
Liste des participants (au 29/04/2022)
- Abdellah Bellahcene
- Florent Blaise
- Séverine Bord
- Guillaume Cornu
- Alexandre Dehne Garcia
- Sylvain Falala
- Thierry Faure
- Laurent Georges
- Marie Grosdidier
- Thierry Hoch
- Loïc Houde
- Jeremy Jachacz
- Aminah KELIET
- Sylvain Lanoe
- Lionel Lellouche
- Mikael Loaec
- Jean-Baptiste Louvet
- Elise Maigne
- Eric Maldonado
- Marine Marjou
- Lucie Michel
- Facundo Muñoz
- Adler Moukouboulou
- Guita Niang
- Marcellino Palerme
- Jean-François Rey
- Isabelle Sanchez
- Hervé Richard
- Luke Riley
- Emily Walker
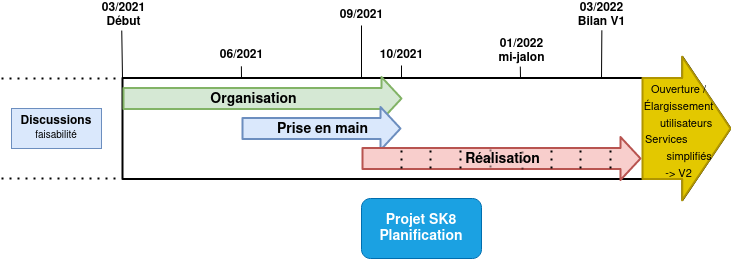
Roadmap
La version 1, prévu pour Avril 2022, se veut être une première brique répondant à la satisfaction des besoins des diffèrentes plateformes d’épidémiosurveillances pour leurs applications. Elle pourra permettre dans une version future de pouvoir proposer une offre à l’echelle de l’institut INRAE.
Planification simplifié du projet.